 |
竹村 和浩(たけむら かずひろ) |
東京工業大学 生命理工学院 北尾研究室 |
研究紹介
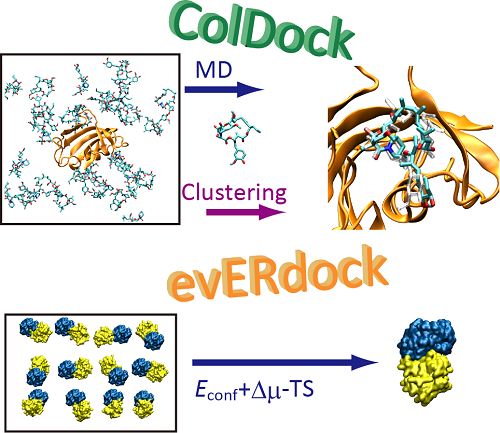
蛋白質は、他の蛋白質やリガンドとの相互作用を通じて機能を発現する為、蛋白質の機能を理解するには複合体の構造や相互作用を正しく理解する必要があります。複合体の構造決定は実験的に決定することが困難な場合が多く、計算による予測が大きく期待されています。「サブ課題A:ポスト「京」でのMD高度化とアルゴリズム深化」では、MD計算を利用し蛋白質複合体構造手法の改善や開発を行っています。蛋白質/蛋白質複合体構造予測に関しては、エネルギー表示法を用いて高精度で複合体モデルの評価を行う手法evERdock(evaluation with Energy Representation method for docking model)を開発しました[2][5][7]
。また、高濃度条件を利用することにより、単純な方法ながら高精度で構造予測が可能である手法ColDock (Concentrated ligand Docking)を開発し[1]、複数の蛋白質/リガンド複合体構造予測に成功しています。現在、開発した手法の効率性や精度向上を行っています。
※[1][2][5][7]は主要論文を参考
■研究分野
・生物物理
■研究課題
蛋白質複合体の構造予測/相互作用解析
■研究キーワード
MD、ドッキング、複合体構造予測、結合自由エネルギー
主要論文・活動実績
■主要論文[1] ColDock: Concentrated Ligand Docking with All-Atom Molecular Dynamics Simulation, K. Takemura, C. Sato, and A. Kitao, J. Phys. Chem. B, DOI: 10.1021/acs.jpcb.8b02756
[2] Binding free energy analysis of protein-protein docking model structures by evERdock, K. Takemura, N. Matubayasi, and A. Kitao, J. Chem. Phys., 148, 105101-1-10, (2018), DOI: 10.1063/1.5019864
[3] Salt bridge formation between the I-BAR domain and lipids increases lipid density and membrane curvature, K Takemura, K Hanawa-Suetsugu, S Suetsugu, A Kitao, Scientific reports 7, 6808 (2017) ,DOI: 10.1038/s41598-017-06334-5
[4] TRPV4 channel activity is modulated by direct interaction of the ankyrin domain to PI(4,5)P2. N. Takahashi, S. Hamada-Nakahara, Y. Itoh, K. Takemura, A. Shimada, Y. Ueda, M. Kitamata, R. Matsuoka, K. Hanawa-Suetsugu, Y. Senju, M. X. Mori, S. Kiyonaka, D. Kohda, A. Kitao, Y. Mori and S. Suetsugu,
Nature Comm. 5, 4994 (2014),DOI: 10.1038/ncomms5994
[5] Free-energy analysis of lysozyme-triNAG binding modes with all-atom molecular dynamics simulation combined with the solution theory in the energy representation. K. Takemura, R.R. Burri, T. Ishikawa, T. Ishikura, S. Sakuraba, N. Matubayasi, K. Kuwata, A. Kitao Chem. Phys. Lett. 559, 94-98 (2013),DOI: 10.1016/j.sbi.2016.10.005
[6] Water Model Tuning for Improved Reproduction of Rotational Diffusion and NMR Spectral Density K. Takemura and A. Kitao J. Phys. Chem. B 116, 6279 (2012),DOI: 10.1021/jp301100g
[7] Evaluation of protein-protein docking model structures using all-atom molecular dynamics simulations combined with the solution theory in the energy representation. K. Takemura, H. Guo, S. Sakuraba, N. Matubayasi, and A. Kitao,
J. Chem. Phys. 137, 215105-1 ~ 215105-10 (2012),DOI:
10.1063/1.4768901
[8] Effects of Water Model and Simulation Box Size on Protein Diffusional Motions
K. Takemura and A. Kitao J. Phys. Chem. B, ACS Publications, 111, 11870-11872 (2007),DOI: 10.1021/jp0756247
[9] Motional coherency in chain dynamics of polybutadiene studied by molecular dynamics simulations K. Takemura, H. Furuya, and T. Kanaya Polymer, 47, 5973 – 5978 (2006),DOI: 10.1016/j.polymer.2006.06.004
■ポスト京研究課題内容の講演
1. An accurate evaluation of protein-protein complexmodels and a simple prediction of protein-ligand complex structures, Kazuhiro Takemura, “Advance in Structural Biology and Beyond (NTHU)”, Hsinchu, Taiwan (2018 Jun).
2. エネルギー表示溶液理論を用いた蛋白質-蛋白質複合体構造予測 竹村 和浩, 松林 伸幸, 北尾 彰朗 “ポスト「京」重点課題1 生体分子システムの機能制御による革新的創薬基盤の構築 共催 ハイパフォーマンス・コンピューティング(HPC)による次世代創薬計算技術”, 日本生物物理学会第55回年会, 熊本, 2017年9月
自己紹介
■経歴
2006年3月 東京工業大学大学院 理工学研究科 有機・高分子物質専攻博士課程修了 博士(工学)
2006年4月 東京大学 分子細胞生物学研究所 産学官連携研究員
2008年4月 分子科学研究所 理論・計算分子科学研究領域 専門研究職員
2011年4月 東京大学 分子細胞生物学研究所 特任研究員
2018年4月 東京工業大学 生命理工学院 特任研究員
■所属学会
・高分子学会(2001年-)
・日本蛋白質科学会(2007年-)
・日本生物物理学会(2007年-)
■研究者になったきっかけとその魅力は?
博士課程終了時、研究は好きだったがまだ自分が納得のいくような研究成果が得られておらず、研究を続けたかったので、研究者になる道を選びました。研究者になってみると自分のまわりには自分より優秀な人がたくさんいて、そんな人たちが長年研究を続けてもまだわからないことがたくさんあります。自分も研究を進めるほどわからないことが増える一方ですが、それを追求していくことが研究のおもしろいところでもあります。
■研究は山あり、谷あり
簡単に出来ると思ったことでつまずいたり、一方、予想外に上手くいくことがよくあります。簡単に出来ると予想していたものほど解決策が思いつかず、研究成果を上げるのに苦労します。つまずいた後でも、自分で解決策を考え、予想通りに行くと喜びは大きいです。
■研究で大切にしていることは?
 |
プレゼンやディスカッション、教育等を含め周囲との適度のコミュニケーションは大切にしています。リラックスした時の研究仲間とのコミュニケーションも大切なひと時です。 (写真右:本人、左:研究仲間の横浜市立大・浴本 亨さん) |
■研究以外の楽しみは?
珈琲が好きなので、珈琲器具にこだわって、じっくり、ゆっくり珈琲タイムを満喫しています。
 |
 |
 |
|
|---|---|---|---|
| 愛用している
珈琲器具達 |
一杯、一杯丁寧にドリップして味わいます。 |
(2018/8/31記録)
他の研究者も見る


