蛋白質・DNA複合系の高精度粗視化モデリングに向けて
【報告日】2016/9/9 ポスト「京」重点課題1 第1回ワークショップ  |
サブ課題A 高田 彰二(京都大学大学院理学研究科) |
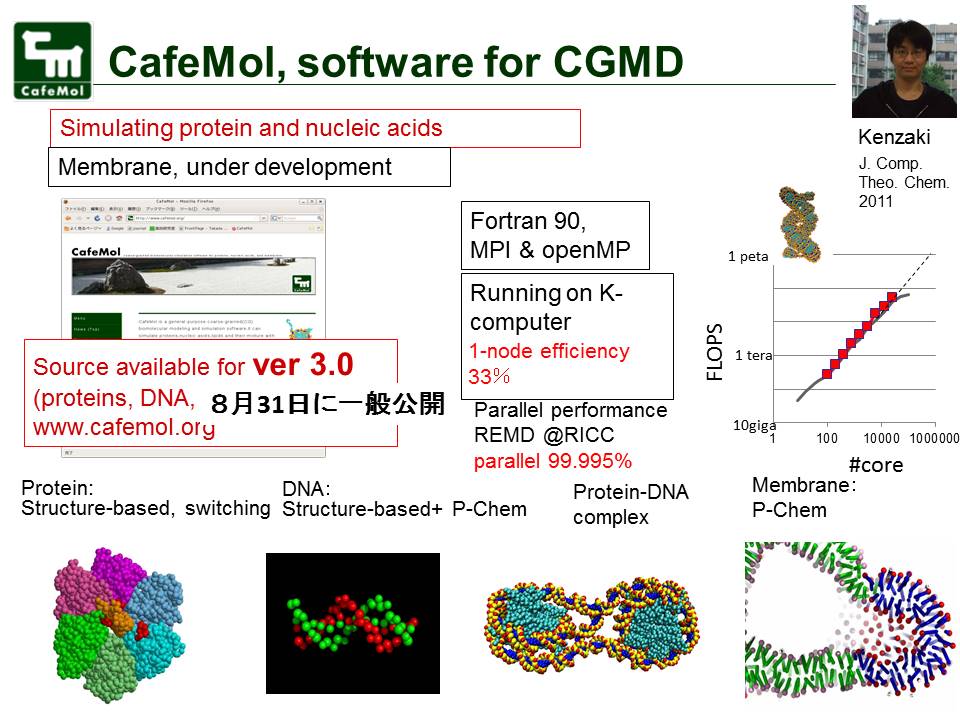
| 我々が進めている粗視化分子シミュレーションの技術的進捗と、細胞核分子モデリング への応用について報告する。まず、粗視化シミュレーションソフトウエアCafeMolのバージ ョン3.0を8月末に一般公開した。今回、新しいDNAモデルが実装され、従来よりかなり高精度で、配列依存的なDNA物性を再現できるようになった。粗視化モデルから全原子モデルを再構築するツールも概ね完成してきた。また、Protein Binding Microarray等の蛋白質DNA相互作用の高スループット実験データを分子シミュレーションに取り込む方法を開発した。応用計算では、ヌクレオソームと転写因子の競合過程等において興味深い現象をシミュレーション中で見出している。 | |
 |
■関連リンク
・研究者情報 高田 彰二
・京都大学理学研究科 生物物理学教室 理論生物物理分科 高田研究室
・粗視化シミュレーションソフトウエアCafeMolのバージ ョン3.0を一般公開(2016/8/31)


